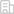2023年8月21日,北京大學生物醫學前沿創新中心(BIOPIC)張澤民課題組聯合中國科學技術大學彭慧課題組,在國際期刊Cell上發表了題為A Pan-Cancer Single Cell Panorama of Human Natural Killer Cells的研究論文。該研究以生物信息學數據整合為支撐,通過收集大規模單細胞轉錄組測序數據,系統刻畫了自然殺傷(NK)細胞在不同癌癥類型和組織之間的異質性,發現了腫瘤微環境(TME)特異富集、殺傷功能異常的NK細胞亞類,揭示了NK細胞與微環境中其他組分的潛在調控關系(圖1)。

圖1. NK細胞單細胞圖譜的圖摘要
近些年來,通過生物信息學方法賦能新技術,解讀復雜現象,已經成為探索和解決重要科學問題的強大助力。與此同時,國內外研究團隊已積累了海量的TME單細胞轉錄組測序數據,系統性整合這些數據資源從而充分挖掘有價值的信息是當前階段研究的重要問題1。但是這些數據的質量參差不齊,來源復雜,測序平臺多樣,因此如何利用數據發現新規律是生物信息遇到的重要挑戰。結合單細胞測序和先進的生物信息數據分析策略,張澤民課題組已成功揭示了多種腫瘤特異的細胞類型2-4。張澤民課題組前期的研究證明了在癌細胞之外,不同腫瘤的免疫微環境存在與患者預后和治療響應相關的穩定的差異,提出了新的腫瘤異質性研究方向。不同的癌癥類型是否有共同的免疫功能異常特征?相同的免疫組分是否在不同組織和條件下保持功能的一致?不同類型的癌癥、不同的患者為什么會對免疫治療產生差異的響應?這些問題的解決將會極大推動腫瘤免疫治療的發展。
本研究聚焦TME中與T細胞功能相似、但具有更直接的識別和殺傷癌細胞能力的NK細胞5。由于腫瘤浸潤的NK細胞在TME的免疫細胞組分中占比稀少,先前的單一研究對其類群分布、功能特性等均缺乏深入、系統性分析。通過生物信息學整合建立大規模NK細胞組學數據資源是突破這一難點的關鍵技術手段。研究者收集整理了大量公開的單細胞轉錄組數據,涉及24種癌癥類型,包括了來自716名患者和47名健康對照的1223個樣本的NK細胞單細胞表達譜數據。研究者克服了數據整合的多項難點,包括高精度分離NK細胞、去除復雜的批次效應等,首次在泛癌水平系統地鑒定到了5類CD56brightCD16lo和9類CD56dimCD16hi NK細胞亞群,并詳細地刻畫了各類群的表型和功能多樣性(圖2)。
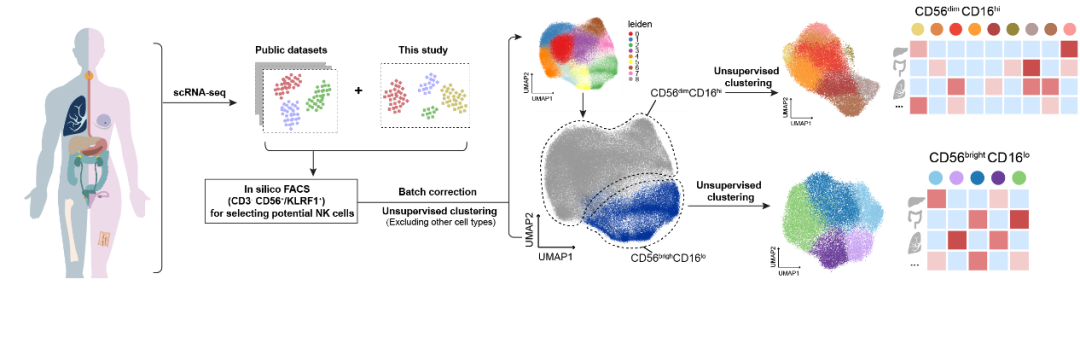
圖2. 課題設計及數據處理主要框架
基于整合的數據資源,研究人員發現NK細胞的亞群組成在不同癌癥類型間表現出了明顯的偏好性,腫瘤、癌旁組織和外周血中的NK細胞亞類分布也具有明顯的差異。通過轉錄組定量分析,敏感性和特異性檢驗等生物信息技術篩選,研究者發現RGS1特異高表達在非血液來源的NK細胞上。相比于經典的組織駐留特征基因,RGS1在轉錄組水平表現出更高的特異性和靈敏度,并且在泛癌種水平上都具有優秀的性能。該發現為后續進一步研究NK細胞的組織浸潤提供了新的研究方向,也進一步突顯了通過整合數據分析研究關鍵生物學問題的可行性。
進一步關注腫瘤微環境,研究者發現腫瘤組織中高度富集一群DNAJB1+ CD56dimCD16hi NK細胞。數據分析發現該群細胞具有功能失調的表型,包括殺傷性下降、抑制受體上升、高表達應激反應相關蛋白等,因此研究者將這一群細胞命名為腫瘤相關NK細胞(Tumor-associated NK cells, TaNK cells)。與經典認知的NK細胞的更高豐度有益于腫瘤患者的生存狀態不同,研究者發現TaNK細胞的富集與多種癌癥類型的不良預后、及免疫治療的耐藥顯著相關。這些發現表明了TaNK細胞具有重要的生物學和臨床應用價值,為后續開發NK細胞相關的免疫治療方法提供了新的思路。除此之外,研究者發現髓系細胞尤其是LAMP3+ DCs是NK細胞的重要調控因子。進一步的生物信息分析結合空間分布定量發現,靠近LAMP3+ DCs的NK細胞表現出更低的殺傷活性,這說明LAMP3+ DCs在腫瘤免疫微環境中對NK細胞功能有著潛在的異常調節作用。
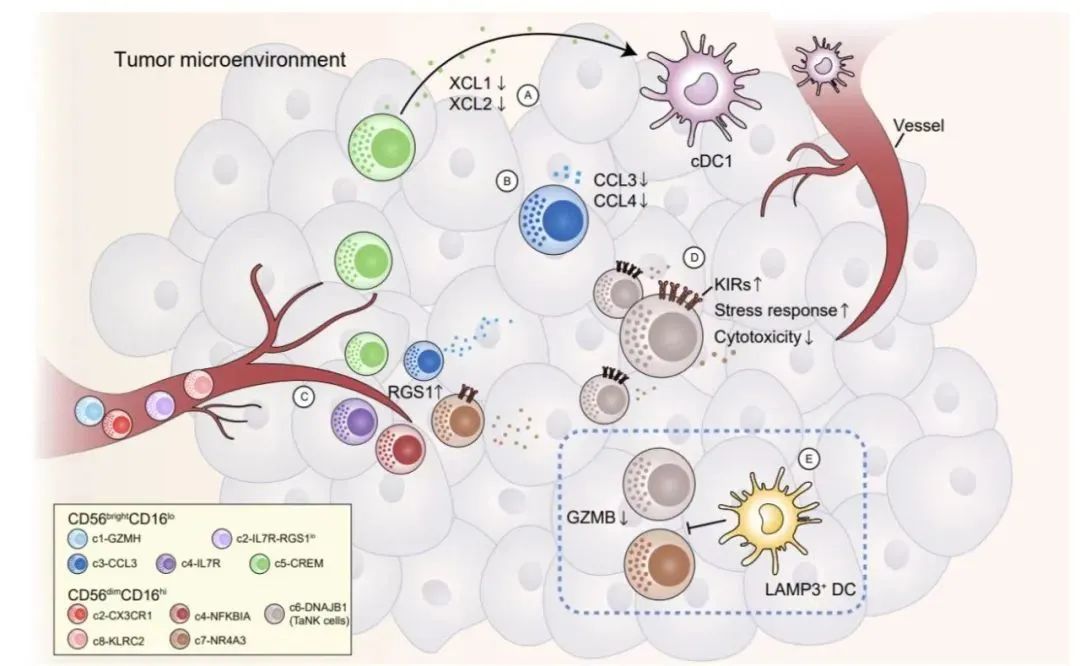
圖3. 研究的主要發現
綜上所述,這一工作創新性地整合利用大規模單細胞數據,揭示了NK細胞中的基因表達模式轉變,捕捉了腫瘤免疫微環境的NK細胞亞群組分變化,為未來通過更全面的整合分析手段探索新的生物標志物和治療靶點提供了助力,也為藥物研發提供更準確、更全面的數據支持。
參考文獻:
1.Stuart, T., and Satija, R. (2019). Integrative single-cell analysis. Nature Reviews Genetics 20, 257-272. 10.1038/s41576-019-0093-7.
2.Zhang, Q., He, Y., Luo, N., Patel, S.J., Han, Y., Gao, R., Modak, M., Carotta, S., Haslinger, C., Kind, D., et al. (2019). Landscape and Dynamics of Single Immune Cells in Hepatocellular Carcinoma. Cell 179, 829-845.e820.https://doi.org/10.1016/j.cell.2019.10.003.
3.Zheng, L., Qin, S., Si, W., Wang, A., Xing, B., Gao, R., Ren, X., Wang, L., Wu, X., Zhang, J., et al. (2021). Pan-cancer single-cell landscape of tumor-infiltrating T cells. Science 374, abe6474. 10.1126/science.abe6474.
4.Zhang, Y., Chen, H., Mo, H., Hu, X., Gao, R., Zhao, Y., Liu, B., Niu, L., Sun, X., Yu, X., et al. (2021). Single-cell analyses reveal key immune cell subsets associated with response to PD-L1 blockade in triple-negative breast cancer. Cancer Cell 39, 1578-1593.e1578.https://doi.org/10.1016/j.ccell.2021.09.010.
5.Myers, J.A., and Miller, J.S. (2021). Exploring the NK cell platform for cancer immunotherapy. Nature Reviews Clinical Oncology 18, 85-100. 10.1038/s41571-020-0426-7.
鏈接:https://authors.elsevier.com/sd/article/S0092-8674(23)00849-8