近日,國際權威期刊《自然·通訊》(Nature Communications)在線發表了中南大學計算機學院李敏教授團隊的最新研究成果“DPFunc:利用結構域引導的結構信息進行蛋白質功能精準預測(DPFunc: Accurately predicting protein function via deep learning with domain-guided structure information)”。該研究提出了一種基于深度學習的新方法DPFunc,有效整合蛋白質序列和結構信息,用于精準預測蛋白質功能。中南大學計算機學院王文康博士為第一作者,計算機學院的李敏教授為論文通訊作者,中南大學為第一署名單位。
蛋白質是生命活動的基本執行單元,精確解析蛋白質功能對于理解生物學機制和疾病治療至關重要。隨著高通量測序技術的快速發展,蛋白質序列數據的規模呈爆炸性增長,但已有的蛋白質功能注釋仍遠遠滯后于數據積累速度。目前,僅有不到1%的蛋白質序列被系統性地注釋了功能信息,這極大地限制了對蛋白質功能的深入理解和相關研究的推進。傳統的實驗方法雖然精準,但成本高、周期長,難以適應大規模蛋白質功能注釋的需求。針對這一挑戰,計算方法成為彌補實驗注釋不足的重要手段。然而,現有蛋白質功能預測方法普遍缺乏可解釋性,難以揭示蛋白質結構與功能之間的關系,導致預測結果的準確性和解釋性有限。尤其是在面對低同源性蛋白或新發現的蛋白質時,這些方法的預測效果大打折扣。
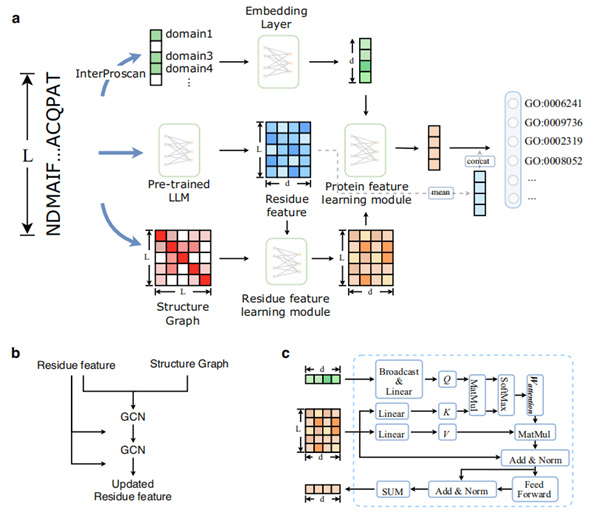
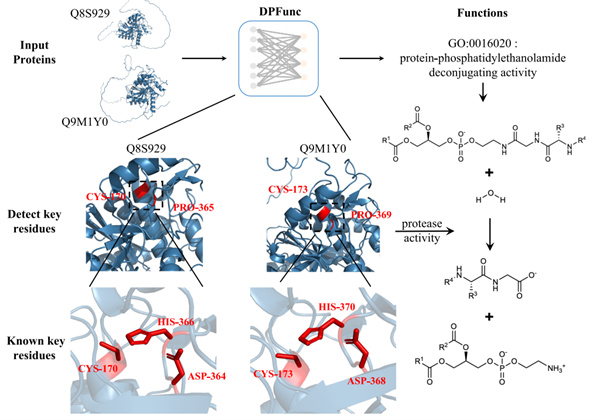
針對這一挑戰,李敏團隊創新性地提出DPFunc方法,通過融合蛋白質序列信息和結構域引導的結構特征,實現了蛋白質功能的精準預測。DPFunc能夠識別蛋白質結構中與功能密切相關的重要區域,在多項基準測試中表現優異,顯著超越現有的最先進方法。實驗結果表明,DPFunc在蛋白質功能預測、關鍵殘基識別和稀有功能檢測等方面表現尤為突出。特別是在蛋白質結構功能關系分析中,DPFunc能夠準確識別關鍵活性位點。例如,在半胱氨酸蛋白酶Q9M1Y0和Q8S929中,DPFunc成功檢測到與功能密切相關的活性位點,表現出出色的解釋性。此外,DPFunc在細菌功能注釋中也展示出強大的潛力,在多種情況下顯著優于其他預測方法,為大規模蛋白質功能預測和疾病機制研究提供了有力工具。
此次研究不僅推動了蛋白質功能預測領域的技術進步,也為理解蛋白質結構與功能的關系提供了新思路。未來,研究團隊將進一步引入更多與蛋白質結構相關的生物學知識,推動DPFunc在動態功能預測和多功能關聯研究中的應用拓展。