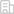東北師范大學生命科學學院張鏵坤教授團隊在植物分子生物學領域取得重要突破。RNA結構和染色質高級構象是國際研究領域的前沿熱點,因其在基因表達調控中的關鍵作用而備受關注。特定的RNA二級結構影響RNA的穩定性、剪接、轉運和翻譯效率,而染色質的三維結構通過環狀結構和拓撲關聯域等特征,影響基因與調控元件的相互作用。這些機制協同作用,使植物在發育和環境響應中實現精確的基因表達調控。2024年,該團隊圍繞“植物體內RNA結構與染色質精細構象與基因表達”問題進行了系統研究,并在國際頂級期刊Nature Machine Intelligence和Nature Communications上連續發表三篇論文,推動了該領域的前沿探索。
張鏵坤教授團隊與英國約翰英納斯中心丁一倞教授團隊合作,首次利用CAP-C技術繪制了高分辨率染色質互作圖譜,揭示了染色質構象與轉錄動態之間的密切關系,尤其是在植物冷脅迫條件下的基因表達調控機制,這一成果為深入理解植物基因表達調控提供了新的思路。相關研究成果以“A fine-scale Arabidopsis chromatin landscape reveals chromatin conformation-associated transcriptional dynamics”為題,發表于國際頂級期刊Nature Communications(2024,15,3253);第一作者為我校生命科學學院師資博士后董芊里博士,英國約翰英納斯中心張月瑩博士、中國科學院遺傳與發育生物學研究所王震博士和芝加哥大學劉沁哲博士,通訊作者為張鏵坤教授和丁一倞教授。
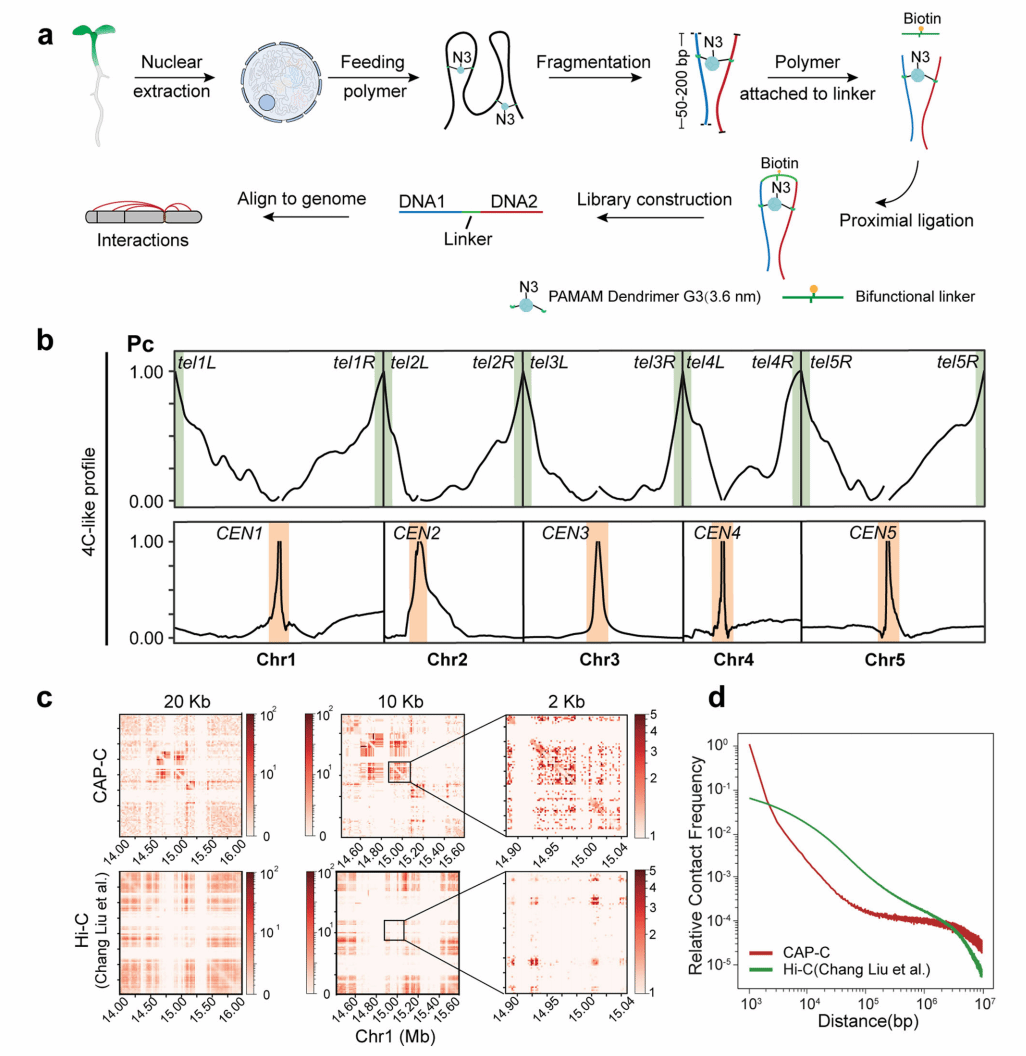
圖1:CAP-C用于擬南芥高分辨率染色質構象
在后續的研究中,團隊在小麥體內RNA結構與穩定性研究中取得重要進展,揭示了小麥mRNA降解主要受3’UTR的RNA結構影響,強調了非編碼區域在基因表達中的重要性,并為小麥馴化和遺傳改良提供了思路。相關研究成果以“Unveiling RNA Structure-mediated Regulations of RNA Stability in Wheat”為題,發表于國際頂級期刊Nature Communications(2024,15,10042);第一作者為我校生命科學學院博士研究生吳海丹,英國約翰英納斯中心于昊澎博士、張月瑩博士和楊畢波博士;通訊作者為張鏵坤教授、劉寶教授和丁一倞教授。
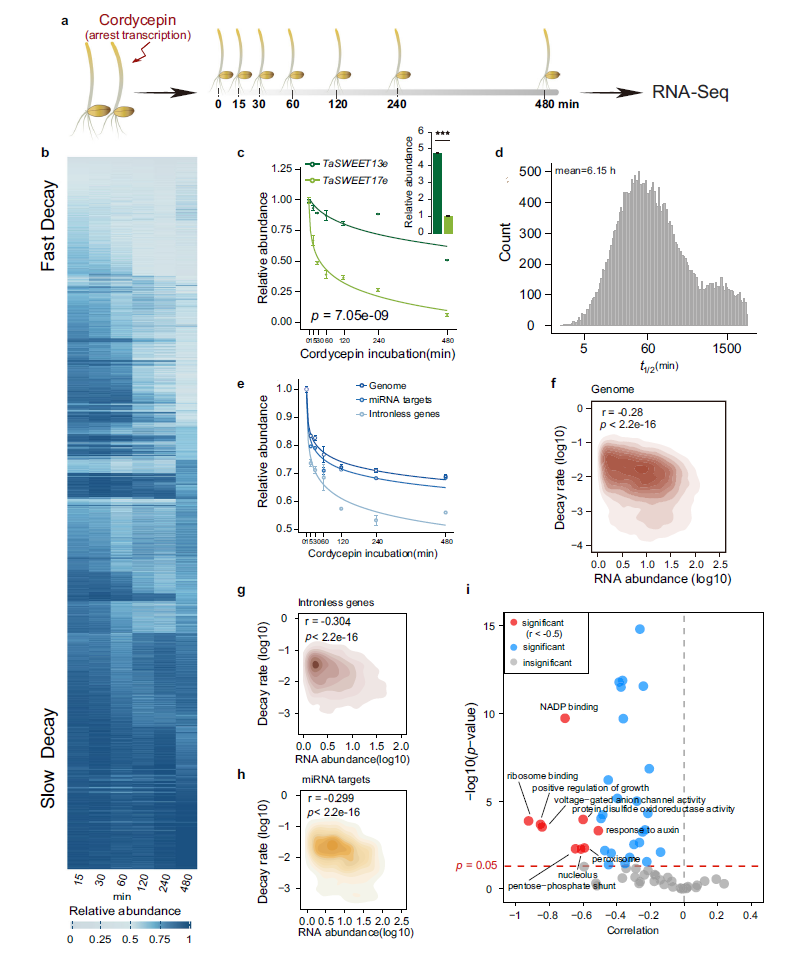
圖2:異源四倍體小麥的mRNA降解圖譜
此外,團隊還關注AI工具在RNA結構預測中的應用,利用PlantRNA-FM模型,團隊整合大規模植物特異數據,成功識別出功能相關的RNA結構基序,為理解植物生長和應激反應背后的分子機制提供了新視角。PlantRNA-FM不僅在植物RNA注釋預測等任務中表現優異,還通過揭示RNA序列和結構的復雜調控網絡,為植物科學研究與應用生物技術提供了強大工具。相關研究成果以“An Interpretable RNA Foundation Model for Exploration of Functional RNA Motifs in Plants”為題,發表于國際頂級期刊Nature Machine Intelligence(2024,in press);第一作者為我校生命科學學院博士研究生孫文青,英國約翰英納斯中心于昊澎博士、英國埃克塞特大學楊恒和英國約翰英納斯中心閆宗運博士;通訊作者為張鏵坤教授、英國約翰英納斯中心丁一倞教授和埃克塞特大學李柯教授。
圖3:PlantRNA-FM模型設計原理圖
近5年來,張鏵坤教授團隊在植物體內RNA結構及染色質構象對基因表達調控的作用方向開展了系統性研究;先后在Nature Machine Intelligence、Nature Communications、Genome Biology、PNAS、The Plant Cell、New Phytologist等國際知名期刊上發表論文30余篇,獲得多項國家和國際合作項目的支持,展現了我校在植物分子生物學領域的科研實力和國際影響力。這些突破性成果不僅為植物科學基礎研究提供了新的工具和方法,也為作物改良和農業發展開辟了新的方向。
相關文章鏈接:
https://www.nature.com/articles/s42256-024-00946-z
https://www.nature.com/articles/s41467-024-54172-7
https://www.nature.com/articles/s41467-024-47678-7